Archivo Europeo de Nucleótidos
| European Nucleotide Archive (ENA) | ||
|---|---|---|
| Parte de ELIXIR EMBL-EBI Node | ||
| Información general | ||
| Tipo de programa | base de datos biológica | |
| Desarrollador | Instituto Europeo de Bioinformática | |
| Lanzamiento inicial | Abril 1982 | |
| Licencia | Irrestricta | |
| Asistencia técnica | ||
| Información sobre secuencia de ácidos nucleicos, proyecto genoma, secuenciación de ADN y secuenciador de ADN, preparación de muestras (química analítica), otros registros relacionados. | ||
| Enlaces | ||
El Archivo Europeo de Nucleótidos (ENA, por sus siglas en inglés) es un repositorio que proporciona acceso libre y sin restricciones a secuencias anotadas de ADN y ARN. También almacena información complementaria como procedimientos experimentales, detalles de ensamblaje de secuencias y otros metadatos relacionados con proyectos de secuenciación.[1] El archivo está compuesto por tres bases de datos principales: el Archivo de Lecturas de Secuencias, el Archivo de Trazas y la Base de Datos de Secuencias de Nucleótidos EMBL (también conocida como EMBL-Bank).[2] El ENA es producido y mantenido por el Instituto Europeo de Bioinformática (EBI) y es miembro de la Colaboración Internacional de Bases de Datos de Secuencias de Nucleótidos (INSDC) junto con el Banco de Datos de ADN de Japón y GenBank.
El ENA evolucionó a partir de la Biblioteca de Datos EMBL, que se lanzó en 1982 como el primer recurso internacionalmente apoyado para datos de secuencias de nucleótidos.[3] A principios de 2012, el ENA y otras bases de datos miembros de INSDC contenían cada una los genomas completos de 5,682 organismos y datos de secuencias para casi 700,000.[4] Además, el volumen de datos está aumentando exponencialmente con un tiempo de duplicación de aproximadamente 10 meses.[5]
Historia
El Archivo Europeo de Nucleótidos tuvo su origen en bases de datos separadas, la más antigua de las cuales fue la Biblioteca de Datos EMBL, establecida en octubre de 1980 en el Laboratorio Europeo de Biología Molecular (EMBL), Heidelberg.[3] La primera publicación de esta base de datos se realizó en abril de 1982 y contenía un total de 568 entradas separadas que consistían en alrededor de 500,000 par de bases.[6] En 1984, refiriéndose a la Biblioteca de Datos EMBL, Kneale y Kennard señalaron que «hace algunos años era evidente que una gran base de datos computarizada de secuencias sería esencial para la investigación en Biología Molecular».[6]

A pesar de que el método de distribución principal en ese momento era mediante cinta magnética, para 1987, se estimaba que la Biblioteca de Datos EMBL era utilizada por unos 10,000 científicos a nivel internacional.[7] Ese mismo año, se introdujo el Servidor de Archivos EMBL para distribuir registros de la base de datos a través de BITNET, EARN y el Internet primitivo.[8] En mayo de 1988, la revista Nucleic Acids Research introdujo una política que establecía que «los manuscritos enviados a [Nucleic Acids Research] que contengan o discutan datos de secuencias deben ir acompañados de evidencia de que los datos han sido depositados en la Biblioteca de Datos EMBL».[9]

Durante la década de 1990, la Biblioteca de Datos EMBL fue renombrada como Base de Datos de Secuencias de Nucleótidos EMBL[10] y fue trasladada formalmente al Instituto Europeo de Bioinformática (EBI) desde Heidelberg.[11] En 2003, la Base de Datos de Secuencias de Nucleótidos se amplió con la adición del Archivo de Versiones de Secuencias (SVA), que mantiene registros de todas las entradas actuales y anteriores en la base de datos.[1] Un año después, en junio de 2004, se eliminaron los límites en la longitud máxima de secuencia para cada registro (entonces 350 kilobases), permitiendo que las secuencias de genomas completos se almacenaran como una sola entrada en la base de datos.[12]
Tras la adopción de la secuenciación de Sanger, el Instituto Sanger Wellcome Trust (entonces conocido como The Sanger Centre) comenzó a catalogar lecturas de secuencias junto con información de calidad en una base de datos llamada Archivo de Trazas.[13] El Archivo de Trazas creció sustancialmente con la comercialización de tecnologías de secuenciación paralela de alto rendimiento por empresas como Roche e Illumina.[14] En 2008, el EBI combinó el Archivo de Trazas, la Base de Datos de Secuencias de Nucleótidos EMBL (ahora también conocida como EMBL-Bank)[2] y un nuevo Archivo de Lecturas de Secuencias (o Short Read Archive, SRA) para formar el ENA, con el objetivo de proporcionar un archivo de secuencias de nucleótidos exhaustivo.[13] Como miembro de la Colaboración Internacional de Bases de Datos de Secuencias de Nucleótidos, el ENA intercambia datos diariamente con el Banco de Datos de ADN de Japón y GenBank.[15]
Base de Datos de Secuencias de Nucleótidos EMBL
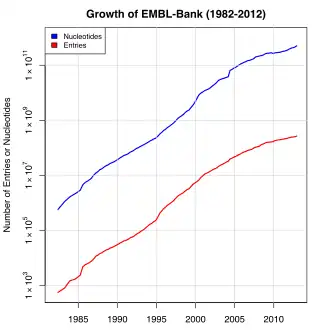
La Base de Datos de Secuencias de Nucleótidos EMBL (también conocida como EMBL-Bank) es la sección del ENA que contiene detalles de ensamblaje del genoma de alto nivel, así como secuencias ensambladas y su anotación funcional.[12][17] EMBL-Bank recibe contribuciones directas de consorcios de genomas y grupos de investigación más pequeños, así como datos de secuencias asociados con patentes.[2][18]
Clases de datos
La Base de Datos de Secuencias de Nucleótidos EMBL soporta una variedad de datos derivados de diferentes fuentes, incluyendo, pero no limitándose a:[19]
- Etiquetas de secuencia expresada con sus datos de muestra asociados.
- Secuencias de nucleótidos generadas a partir de proyectos de secuenciación del genoma completo en diversas etapas de ensamblaje, incluyendo contigs completos y secuencias completamente ensambladas y anotadas.
- Datos relacionados con transcriptómica, como ADN complementario, con anotaciones opcionales.
- Anotaciones nuevas o extendidas de secuencias codificantes existentes, por ejemplo, nuevas versiones de secuencias con inicio o terminación corregidos.
Formato EMBL-Bank
La Base de Datos de Secuencias de Nucleótidos EMBL utiliza un formato de archivo plano en texto plano conocido como formato EMBL-Bank.[20] El formato EMBL-Bank utiliza una sintaxis diferente a los registros de DDBJ y GenBank, aunque cada formato utiliza ciertas nomenclaturas estandarizadas, como las taxonomías definidas por la base de datos taxonómica del NCBI. Cada línea de un archivo en formato EMBL comienza con un código de dos letras, como AC para el número de acceso y KW para una lista de palabras clave relevantes al registro; cada registro termina con //.[20]
Archivo de Lecturas de Secuencias
_size_of_the_Sequence_Read_Archive.svg.png)
El ENA opera una instancia del Archivo de Lecturas de Secuencias (SRA), un repositorio de archivo de lecturas de secuencias y análisis destinados a la publicación pública.[23] Originalmente llamado Short Read Archive, el nombre cambió en previsión de que futuras tecnologías de secuenciación pudieran producir lecturas de secuencias más largas.[24] Actualmente, el archivo acepta lecturas de secuencias generadas por plataformas de secuenciación de próxima generación como el Analizador de Genomas de Illumina y ABI SOLiD, así como algunos análisis y alineaciones correspondientes.[25] El SRA opera bajo la guía de la Colaboración Internacional de Bases de Datos de Secuencias de Nucleótidos (INSDC)[23] y es el repositorio de más rápido crecimiento en el ENA.[14]
En 2010, el Archivo de Lecturas de Secuencias representaba aproximadamente el 95% de los datos de par de bases disponibles a través del ENA,[13] abarcando más de 500,000,000,000 de lecturas de secuencias compuestas por más de 60 billones (6×1013) de pares de bases.[23] Casi la mitad de estos datos se depositaron en relación con el Proyecto 1000 Genomas[23] donde los investigadores publicaron sus datos de secuencias en el SRA en tiempo real.[26] En total, hasta septiembre de 2010, el 65% del Archivo de Lecturas de Secuencias era secuencia genómica humana, con otro 16% relacionado con lecturas de secuencias de metagenoma humano.[23]
El formato de datos preferido para los archivos enviados al SRA es el formato BAM, que puede almacenar lecturas alineadas y no alineadas.[23] Internamente, el SRA utiliza el NCBI SRA Toolkit, usado en las tres bases de datos miembros de INSDC, para proporcionar una compresión de datos flexible, acceso a la API y conversión a otros formatos como FASTQ.[22]
Acceso a los datos
Los datos contenidos en el ENA pueden accederse manualmente o mediante programación a través de URL REST a través del navegador ENA. Inicialmente limitado al Archivo de Lecturas de Secuencias,[14] el navegador ENA ahora también proporciona acceso al Archivo de Trazas y EMBL-Bank, permitiendo la recuperación de archivos en una variedad de formatos, incluyendo XML, HTML, FASTA y FASTQ.[13] Los registros individuales pueden accederse utilizando sus números de acceso y otras consultas de texto están habilitadas a través del motor de búsqueda EB-eye.[13] Además, las búsquedas basadas en similitud de secuencias implementadas usando grafos de De Bruijn ofrecen otro método para recuperar registros del ENA.[14]
El ENA es accesible a través de las API SOAP y REST del EBI, que también ofrecen acceso a otras bases de datos alojadas en el EBI, como Ensembl e InterPro.[27]
Almacenamiento
El Archivo Europeo de Nucleótidos maneja grandes volúmenes de datos que representan un desafío significativo de almacenamiento.[5][28] Hasta 2012, los requisitos de almacenamiento del ENA continuaban creciendo exponencialmente, con un tiempo de duplicación de aproximadamente 10 meses.[5] Para gestionar este aumento, el ENA descarta selectivamente datos de plataformas de secuenciación menos valiosos e implementa estrategias avanzadas de compresión.[23][29] El kit de herramientas de compresión basado en referencias CRAM fue desarrollado para ayudar a reducir los requisitos de almacenamiento del ENA.[5][30]
Financiamiento
Actualmente, el ENA es financiado conjuntamente por el Laboratorio Europeo de Biología Molecular, la Comisión Europea y el Wellcome Trust.[13] El marco emergente ELIXIR, coordinado por la directora del EBI Janet Thornton, busca asegurar una infraestructura de financiación europea sostenible para apoyar la disponibilidad continua de bases de datos de ciencias de la vida como el ENA.[29][31][32]
Véase también
Referencias
- ↑ a b Cochrane, G.; Akhtar, R.; Aldebert, P.; Althorpe, N.; Baldwin, A.; Bates, K.; Bhattacharyya, S.; Bonfield, J. et al. (2007). «Priorities for nucleotide trace, sequence and annotation data capture at the Ensembl Trace Archive and the EMBL Nucleotide Sequence Database» [Prioridades para la captura de datos de trazas, secuencias y anotaciones en el Ensembl Trace Archive y la Base de Datos de Secuencias de Nucleótidos EMBL]. Nucleic Acids Research 36 (Base de datos): D5-D12. ISSN 0305-1048. PMC 2238915. PMID 18039715. doi:10.1093/nar/gkm1018. Consultado el 3 de julio de 2025.
- ↑ a b c EMBL-EBI. «EMBL Nucleotide Sequence Database» [Base de Datos de Secuencias de Nucleótidos EMBL]. Consultado el 3 de julio de 2025.
- ↑ a b Hamm, G. H.; Cameron, G. N. (1986). «The EMBL data library» [La biblioteca de datos EMBL]. Nucleic Acids Research 14 (1): 5-9. PMC 339348. PMID 3945550. doi:10.1093/nar/14.1.5. Consultado el 3 de julio de 2025.
- ↑ Cochrane, Guy; Cook, Charles E; Birney, Ewan (2012). «The future of DNA sequence archiving» [El futuro del archivo de secuencias de ADN]. GigaScience 1 (1): 2. ISSN 2047-217X. PMC 3617450. PMID 23587147. doi:10.1186/2047-217X-1-2. Consultado el 3 de julio de 2025.
- ↑ a b c d Cochrane, G.; Alako, B.; Amid, C.; Bower, L.; Cerdeno-Tarraga, A.; Cleland, I.; Gibson, R.; Goodgame, N. et al. (2012). «Facing growth in the European Nucleotide Archive» [Enfrentando el crecimiento en el Archivo Europeo de Nucleótidos]. Nucleic Acids Research 41 (D1): D30-D35. ISSN 0305-1048. PMC 3531187. PMID 23203883. doi:10.1093/nar/gks1175. Consultado el 3 de julio de 2025.
- ↑ a b Kneale, G.; Kennard, O. (1984). «The EMBL nucleotide sequence data library» [La biblioteca de datos de secuencias de nucleótidos EMBL]. Biochemical Society Transactions 12 (6): 1011-1014. PMID 6530028. doi:10.1042/bst0121011.
- ↑ Cameron, G. N. (1988). «The EMBL data library» [La biblioteca de datos EMBL]. Nucleic Acids Research 16 (5): 1865-1867. PMC 338182. PMID 3353226. doi:10.1093/nar/16.5.1865. Consultado el 3 de julio de 2025.
- ↑ Fuchs, R.; Stoehr, P.; Rice, P.; Omond, R.; Cameron, G. (1990). «New services of the EMBL Data Library» [Nuevos servicios de la Biblioteca de Datos EMBL]. Nucleic Acids Research 18 (15): 4319-4323. PMC 331247. PMID 2388823. doi:10.1093/nar/18.15.4319. Consultado el 3 de julio de 2025.
- ↑ Kahn, P.; Hazledine, D. (1988). «NAR's new requirement for data submission to the EMBL data library: Information for authors» [Nuevo requisito de NAR para la presentación de datos a la Biblioteca de Datos EMBL: Información para autores]. Nucleic Acids Research 16 (10): I-IV. PMC 336623. PMID 16617480. Consultado el 3 de julio de 2025.
- ↑ «What is the European Nucleotide Archive?» [¿Qué es el Archivo Europeo de Nucleótidos?]. EMBL-EBI. Consultado el 3 de julio de 2025.
- ↑ Rodriguez-Tomé, P.; Stoehr, P. J.; Cameron, G. N.; Flores, T. P. (1996). «The European Bioinformatics Institute (EBI) databases» [Las bases de datos del Instituto Europeo de Bioinformática (EBI)]. Nucleic Acids Research 24 (1): 6-12. PMC 145572. PMID 8594602. doi:10.1093/nar/24.1.6. Consultado el 3 de julio de 2025.
- ↑ a b Stoesser, G.; Baker, W; Van Den Broek, A; Garcia-Pastor, M; Kanz, C; Kulikova, T; Leinonen, R; Lin, Q et al. (2003). «The EMBL Nucleotide Sequence Database: major new developments» [La Base de Datos de Secuencias de Nucleótidos EMBL: principales nuevos desarrollos]. Nucleic Acids Research 31 (1): 17-22. ISSN 1362-4962. PMC 165468. PMID 12519939. doi:10.1093/nar/gkg021. Consultado el 3 de julio de 2025.
- ↑ a b c d e f «The European Nucleotide Archive» [El Archivo Europeo de Nucleótidos]. Nucleic Acids Res. 39 (Edición de base de datos): D28-31. enero de 2011. PMC 3013801. PMID 20972220. doi:10.1093/nar/gkq967. Consultado el 3 de julio de 2025.
- ↑ a b c d Leinonen, R.; Akhtar, R.; Birney, E.; Bonfield, J.; Bower, L.; Corbett, M.; Cheng, Y.; Demiralp, F. et al. (2009). «Improvements to services at the European Nucleotide Archive» [Mejoras en los servicios del Archivo Europeo de Nucleótidos]. Nucleic Acids Research 38 (Base de datos): D39-D45. ISSN 0305-1048. PMC 2808951. PMID 19906712. doi:10.1093/nar/gkp998. Consultado el 3 de julio de 2025.
- ↑ EMBL-EBI. «About the European Nucleotide Archive» [Acerca del Archivo Europeo de Nucleótidos]. Consultado el 3 de julio de 2025.
- ↑ "EMBL Nucleotide Sequence Database: Release Notes". EMBL-Bank Release Notes 114. EMBL-EBI. Dec 2012. Archivado del original el 2 de enero de 2013. Consultado el 7 de enero de 2013.
- ↑ Amid, C.; Birney, E.; Bower, L.; Cerdeno-Tarraga, A.; Cheng, Y.; Cleland, I.; Faruque, N.; Gibson, R. et al. (2011). «Major submissions tool developments at the European nucleotide archive» [Principales desarrollos de herramientas de presentación en el Archivo Europeo de Nucleótidos]. Nucleic Acids Research 40 (D1): D43-D47. ISSN 0305-1048. PMC 3245037. PMID 22080548. doi:10.1093/nar/gkr946. Consultado el 3 de julio de 2025.
- ↑ Stoesser, G.; Baker, W; Van Den Broek, A; Camon, E; Garcia-Pastor, M; Kanz, C; Kulikova, T; Leinonen, R et al. (2002). «The EMBL Nucleotide Sequence Database» [La Base de Datos de Secuencias de Nucleótidos EMBL]. Nucleic Acids Research 30 (1): 21-26. ISSN 1362-4962. PMC 99098. PMID 11752244. doi:10.1093/n/nar/30.1.21. Consultado el 3 de julio de 2025.
- ↑ «EMBL-Bank data classes» [Clases de datos de EMBL-Bank]. EMBL-EBI. 2012. Consultado el 3 de julio de 2025.
- ↑ a b «EMBL-Bank User Manual (Release 129)» [Manual de usuario de EMBL-Bank (Versión 129)] (Texto plano). EMBL-EBI. septiembre de 2016. Consultado el 3 de julio de 2025.
- ↑ «NCBI SRA Overview» [Resumen del SRA de NCBI]. NCBI. 1 de enero de 2013. Archivado desde el original el 8 de febrero de 2013. Consultado el 3 de julio de 2025.
- ↑ a b Kodama, Y.; Shumway, M.; Leinonen, R. (2011). «The sequence read archive: explosive growth of sequencing data» [El archivo de lecturas de secuencias: crecimiento explosivo de datos de secuenciación]. Nucleic Acids Research 40 (D1): D54-D56. ISSN 0305-1048. PMC 3245110. PMID 22009675. doi:10.1093/nar/gkr854. Consultado el 3 de julio de 2025.
- ↑ a b c d e f g «The sequence read archive». Nucleic Acids Res. 39 (Edición de base de datos): D19-21. enero de 2011. PMC 3013647. PMID 21062823. doi:10.1093/nar/gkq1019. Consultado el 3 de julio de 2025.
- ↑ Ostell, Jim (2009). «NCBI's Sequence Read Archive: A Core Enabling Infrastructure» [El Archivo de Lecturas de Secuencias de NCBI: Una infraestructura habilitadora central]. Bio IT World. Consultado el 3 de julio de 2025.
- ↑ «About the NCBI Sequence Read Archive» [Acerca del Archivo de Lecturas de Secuencias de NCBI]. NCBI. 8 de enero de 2013. Archivado desde el original el 19 de abril de 2013. Consultado el 3 de julio de 2025.
- ↑ Shumway, M.; Cochrane, G.; Sugawara, H. (2009). «Archiving next generation sequencing data» [Archivando datos de secuenciación de próxima generación]. Nucleic Acids Research 38 (Base de datos): D870-D871. ISSN 0305-1048. PMC 2808927. PMID 19965774. doi:10.1093/nar/gkp1078. Consultado el 3 de julio de 2025.
- ↑ Mcwilliam, H.; Valentin, F.; Goujon, M.; Li, W.; Narayanasamy, M.; Martin, J.; Miyar, T.; Lopez, R. (2009). «Web services at the European Bioinformatics Institute-2009» [Servicios web en el Instituto Europeo de Bioinformática-2009]. Nucleic Acids Research 37 (Servidor web): W6-W10. ISSN 0305-1048. PMC 2703973. PMID 19435877. doi:10.1093/nar/gkp302. Consultado el 3 de julio de 2025.
- ↑ Cochrane, G.; Akhtar, R.; Bonfield, J.; Bower, L.; Demiralp, F.; Faruque, N.; Gibson, R.; Hoad, G. et al. (2009). «Petabyte-scale innovations at the European Nucleotide Archive» [Innovaciones a escala de petabytes en el Archivo Europeo de Nucleótidos]. Nucleic Acids Research 37 (Base de datos): D19-D25. ISSN 0305-1048. PMC 2686451. PMID 18978013. doi:10.1093/nar/gkn765. Consultado el 3 de julio de 2025.
- ↑ a b «EMBL-EBI will continue to support the Sequence Read Archive for raw data» [EMBL-EBI continuará apoyando el Archivo de Lecturas de Secuencias para datos sin procesar]. Comunicado de prensa (EMBL-EBI). 16 de febrero de 2011. Archivado desde el original el 15 de mayo de 2011. Consultado el 3 de julio de 2025.
- ↑ Hsi-Yang Fritz, M.; Leinonen, R.; Cochrane, G.; Birney, E. (2011). «Efficient storage of high throughput DNA sequencing data using reference-based compression» [Almacenamiento eficiente de datos de secuenciación de ADN de alto rendimiento usando compresión basada en referencias]. Genome Research 21 (5): 734-740. ISSN 1088-9051. PMC 3083090. PMID 21245279. doi:10.1101/gr.114819.110. Consultado el 3 de julio de 2025.
- ↑ «About ELIXIR» [Acerca de ELIXIR]. ELIXIR. Archivado desde el original el 4 de octubre de 2011. Consultado el 20 de febrero de 2025.
- ↑ Crosswell, Lindsey C.; Thornton, Janet M. (2012). «ELIXIR: a distributed infrastructure for European biological data» [ELIXIR: una infraestructura distribuida para datos biológicos europeos]. Trends in Biotechnology 30 (5): 241-242. ISSN 0167-7799. PMID 22417641. doi:10.1016/j.tibtech.2012.02.002.